plot_covid Intenta graficar automaticamente la base de datos de covid generados por casos()
Uso
plot_covid(
datos_covid,
df_name = "casos",
df_date_index = stringr::str_subset(colnames(datos_covid[df_name][[1]]),
"FECHA|fecha|Fecha"),
df_variable = NULL,
df_covariates = c(),
facet_scale = "free_y",
facet_ncol = 4,
date_break_format = "2 months",
date_labels_format = "%B-%y",
type = c("point", "line", "spline", "area"),
plot_theme = ggplot2::theme(panel.background = ggplot2::element_rect(fill = "white"),
plot.background = ggplot2::element_rect(fill = "white"), axis.text.x =
ggplot2::element_text(angle = 90, hjust = 1), axis.line.x =
ggplot2::element_line(color = "black"), legend.position = "none"),
...
)Argumentos
- datos_covid
(obligatorio) Lista de
tibbles resultante decasos(),cfr(),chr(),positividad()ort()- df_name
(opcional) Nombre de la base de datos dentro de la lista
datos_covid- df_date_index
(opcional) Nombre de la variable que contiene la fecha
- df_variable
(opcional) Nombre de la variable que se va a graficar en el eje y
- df_covariates
(opcional) Covariables para el
facet_wrap(maximo 2)- facet_scale
(opcional) Escala para el
ggplot2::facet_wrap()- facet_ncol
(opcional) Numero de columnas para el
ggplot2::facet_wrap()- date_break_format
(opcional) Breaks para el eje x
ggplot2::scale_x_date()- date_labels_format
(opcional) Formato de fecha para el eje x
ggplot2::scale_x_date()- type
(opcional) Tipo de grafica (
line,area,splineopoint)- plot_theme
(opcional) Tema para el
ggplot2(ejemploggplot2::theme_classic()).- ...
(opcional) Parametros adicionales para
ggformula::geom_spline()en caso de elegirtype="spline"
Ejemplos
# Para el ejemplo usaremos los datos precargados (datosabiertos) pero tu puedes
# correr el ejemplo descargando informacion mas reciente:
datos_covid <- datosabiertos
# Aqui muchos aparecen en cero si usas el default de datosabiertos
# porque la base de datosabiertos tiene muy pocos casos
datos_covid |>
casos(list_name = "casos_for_plot", group_by_entidad = FALSE) |>
plot_covid(df_name = "casos_for_plot")
#> ! `df_variable` no fue especificada. Usaremos la columna n
#> ! `df_covariates` no fue especificada. Usaremos ``
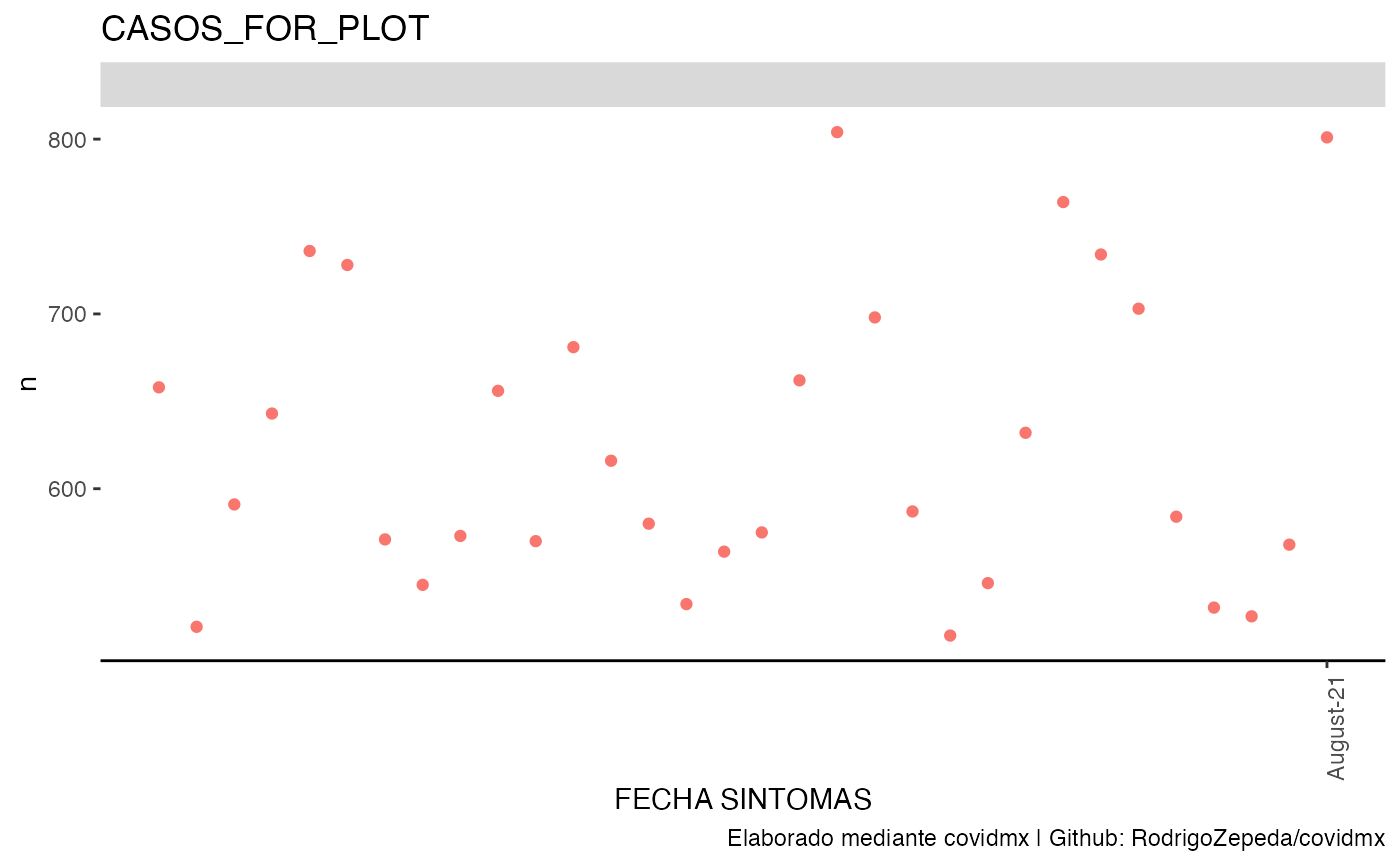 # Grafica de casos nacional
# \donttest{
datos_covid |>
casos(group_by_entidad = FALSE, list_name = "plot_nal") |>
plot_covid(df_name = "plot_nal")
#> ! `df_variable` no fue especificada. Usaremos la columna n
#> ! `df_covariates` no fue especificada. Usaremos ``
# Grafica de casos nacional
# \donttest{
datos_covid |>
casos(group_by_entidad = FALSE, list_name = "plot_nal") |>
plot_covid(df_name = "plot_nal")
#> ! `df_variable` no fue especificada. Usaremos la columna n
#> ! `df_covariates` no fue especificada. Usaremos ``
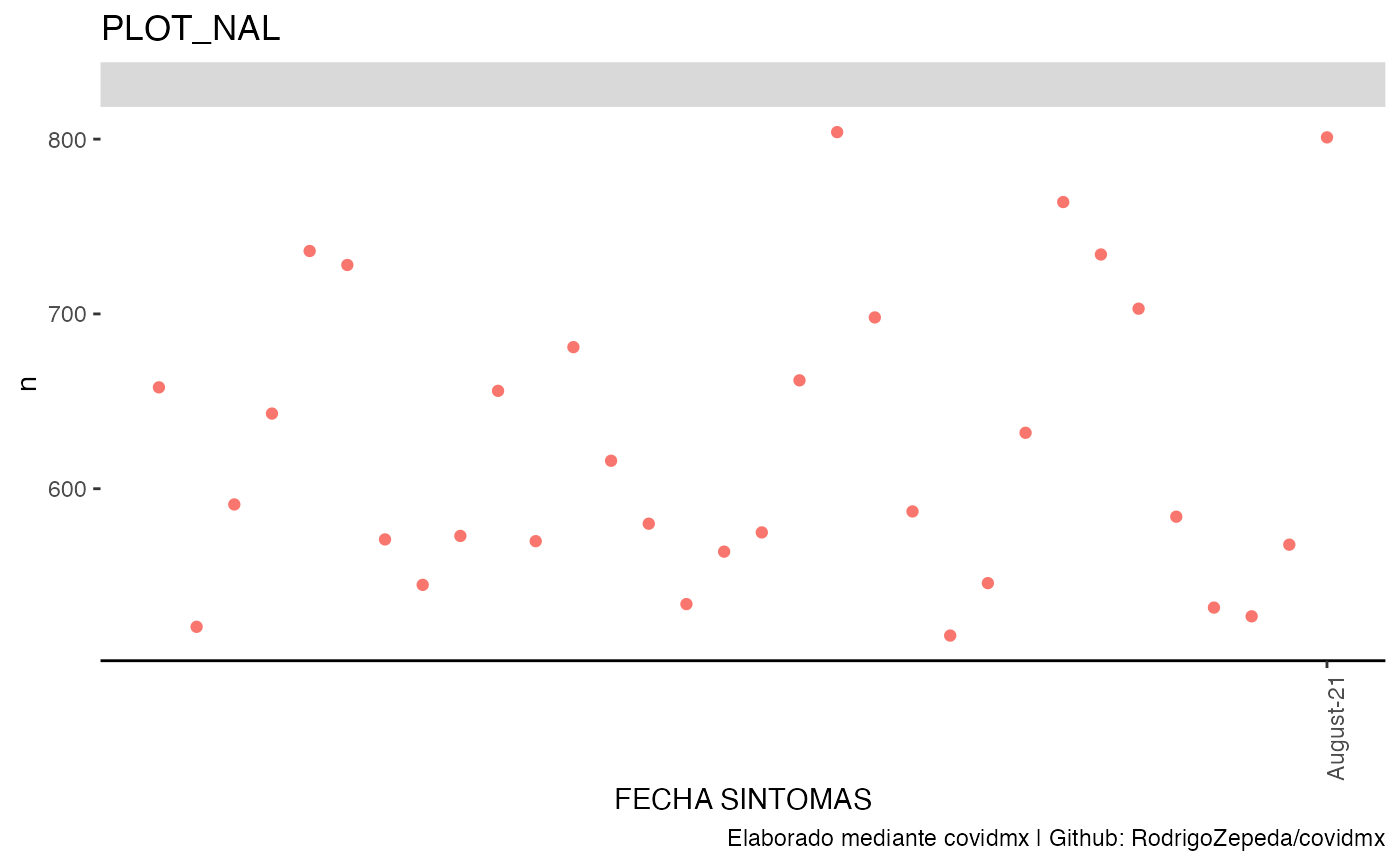 # Ajuste mediante splines
datos_covid |>
casos(group_by_entidad = FALSE, list_name = "spline_nacional") |>
plot_covid(df_name = "spline_nacional", type = "spline", spar = 0.5)
#> ! `df_variable` no fue especificada. Usaremos la columna n
#> ! `df_covariates` no fue especificada. Usaremos ``
# Ajuste mediante splines
datos_covid |>
casos(group_by_entidad = FALSE, list_name = "spline_nacional") |>
plot_covid(df_name = "spline_nacional", type = "spline", spar = 0.5)
#> ! `df_variable` no fue especificada. Usaremos la columna n
#> ! `df_covariates` no fue especificada. Usaremos ``
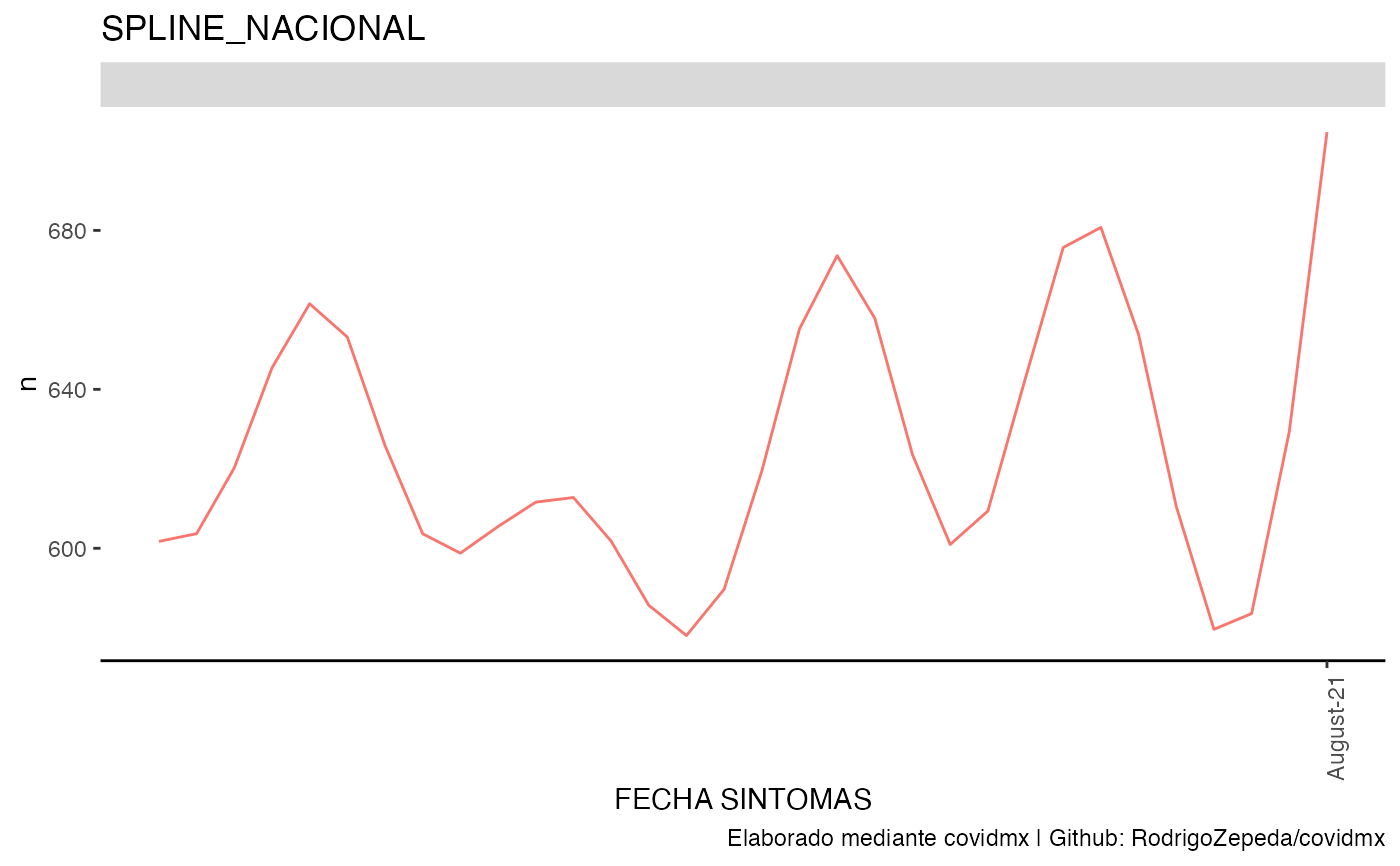 # Graficacion por covariables
# el objeto devuelto es un objeto de ggplot2 al que se le puede dar formato
if (!requireNamespace("ggplot2", quietly = TRUE)) {
datos_covid |>
chr(
group_by_entidad = TRUE, list_name = "plot_nal", .grouping_vars = c("SEXO"),
entidades = c("BAJA CALIFORNIA", "BAJA CALIFORNIA SUR")
) |>
plot_covid(
df_name = "plot_nal",
date_break_format = "1 week",
date_labels_format = "%d/%B/%Y",
df_covariates = c("SEXO", "ENTIDAD_FEDERATIVA"),
type = "area"
) +
ggplot2::ggtitle("Plot nacional")
}
# Puedes tambien primero editar el tibble que usaras por ejemplo poniendo
# los nombres de los sexos
datos_covid <- datos_covid |>
chr(
group_by_entidad = TRUE, list_name = "plot_nal", .grouping_vars = c("SEXO"),
entidades = c("BAJA CALIFORNIA", "BAJA CALIFORNIA SUR")
)
# }
# Finalmente desconectamos
datos_covid$disconnect()
#> ✔ Desconectado
# Graficacion por covariables
# el objeto devuelto es un objeto de ggplot2 al que se le puede dar formato
if (!requireNamespace("ggplot2", quietly = TRUE)) {
datos_covid |>
chr(
group_by_entidad = TRUE, list_name = "plot_nal", .grouping_vars = c("SEXO"),
entidades = c("BAJA CALIFORNIA", "BAJA CALIFORNIA SUR")
) |>
plot_covid(
df_name = "plot_nal",
date_break_format = "1 week",
date_labels_format = "%d/%B/%Y",
df_covariates = c("SEXO", "ENTIDAD_FEDERATIVA"),
type = "area"
) +
ggplot2::ggtitle("Plot nacional")
}
# Puedes tambien primero editar el tibble que usaras por ejemplo poniendo
# los nombres de los sexos
datos_covid <- datos_covid |>
chr(
group_by_entidad = TRUE, list_name = "plot_nal", .grouping_vars = c("SEXO"),
entidades = c("BAJA CALIFORNIA", "BAJA CALIFORNIA SUR")
)
# }
# Finalmente desconectamos
datos_covid$disconnect()
#> ✔ Desconectado